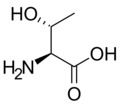
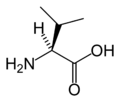
Aminoácido proteinoxénico
Os aminoácidos proteinoxénicos son os aminoácidos que forman parte das proteínas e están codificados no código xenético.[1] Hai 20 aminoácidos codificados de forma normal no código xenético e outros 2 codificados dun xeito especial. Estes últimos son a selenocisteína e a pirrolisina. A estes habería que engadir a N-formilmetionina, un derivado de aminoácido co que se inicia a síntese proteica en bacterias, mitocondrias e cloroplastos, polo que contando este farían un total de 23.
A selenocisteína está codificada polo codón de parada ou stop UGA do código xenético, que normalmente determina o fin da síntese dunha proteína, pero se existe un elemento SECIS no ARNm este codón inserirá selenocisteína na proteína que se está a sintetizar en bacterias, arqueas ou eucariotas. Neste sistema actúan un ARNt e unha aminoacil ARNt sintetase especiais.
A pirrolisina é incorporada nalgunhas arqueas e bacterias (non en eucariotas) durante a síntese normal das proteínas cando o codón UAG, que é un codón de parada de finalización da síntese, é lido como un codón para a pirrolisina debido á actuación dun ARNt especial e unha aminoacil ARNt sintetase especial, e, segundo unha teoría, á presenza dun elemento PYLIS no ARNm, aínda que este elemento parece que non funciona igual nin é tan esencial coma o elemento SECIS para a selenocisteína.[2][3][4]
A N-formilmetionina inicia a síntese proteica en bacterias, mitocondrias e cloroplastos, pero non en arqueas nin nas proteínas eucariotas sintetizadas nos ribosomas do citosol ou retículo endoplasmático rugoso. Está codificada polo codón AUG, que normalmente codifica a metionina. A N-formilmetionina, igual que os demais aminoácidos, ten un ARNt especial que a leva ao codón AUG. Este ARNt carga inicialmente metionina pero despois engádeselle encimaticamente o grupo formilo, orixinando a N-formilmetionina. Normalmente, este aminoácido é eliminado da proteína despois por modificación postraducional, pero non sempre. Tecnicamente a N-formilmetionina é un derivado de aminoácido (xa que o formilo está unido precisamente ao grupo amino) máis que un aminoácido normal e as máis das veces é eliminada da proteína despois da tradución, polo que ás veces non se conta como aminoácido proteinoxénico, pero a súa codificación é, basicamente, coma a doutro aminoácido calquera.
Por tanto, hai en total 22 aminoácidos estándar nos seres vivos e só 21 nos eucariotas (falta a pirrolisina). Serían 23 se contamos a formilmetionina, que nos eucariotas só se utiliza en mitocondrias e cloroplastos e nas bacterias, e moitas veces pouco despois da síntese é eliminada. Todos os demais aminoácidos existentes considéranse non proteinoxénicos.
O termo proteinoxénico significa que xeran ou constrúen as proteínas durante a súa síntese ribosómica, xa que durante a tradución das proteínas se unen uns a outros por enlace peptídico formando os polipéptidos ou proteínas. Polo contrario, os aminoácidos non proteinoxénicos ou non forman parte das proteínas (como a carnitina, GABA, ou L-DOPA), ou son resultado da modificación postraducional dun aminoácido normal (como a hidroxiprolina, que procede da hidroxilación de residuos de prolina nas proteínas), ou incorporados de forma excepcional en substitución doutro aminoácido (como a selenometionina, que pode ocupar o sitio da metionina durante a síntese). Os aminoácidos non proteinoxénicos poden ser incorporados aos péptidos non ribosómicos, que non se sintetizan nos ribosomas.
Hai razóns claras polas que na evolución inicial dos organismos non se incorporaron ás proteínas certos aminoácidos non proteinoxénicos. Os aminoácidos proteinoxénicos están relacionados co conxunto de aminoácidos que nalquel momento inicial podían ser recoñecidos por sistemas de autoaminoacilación de ribozimas.[5] Así, os aminoácidos non proteinoxénicos terían sido excluídos polo éxito evolutivo das formas de vida baseadas nos nucleótidos. Outras razóns son, por exemplo, que a ornitina e a homoserina se ciclan coa estrutura do peptido e fragmentan a proteína facilmente, mentres que outros son tóxicos porque poden ser incorporados por erro ás proteínas, como ocorre coa canavanina, análogo da arxinina.
Os seres humanos non poden sintetizar no seu metabolismo a partir doutros precursores sinxelos os 20 aminoácidos codificados de forma normal no código xenético, senón só 11, polo que os restantes 9 considéranse aminoácidos esenciais, que deben ser tomados nos alimentos da dieta, e son: histidina, isoleucina, leucina, lisina, metionina, fenilalanina, treonina, triptófano, e valina. Algúns aminoácidos só son necesarios en situacións especiais.
Estruturas
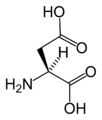
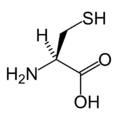
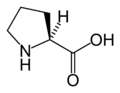
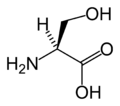
editarAs figuras ilustran as estruturas e indican os nomes e as abreviacións de tres letras e dunha letra dos 21 aminoácidos que están directamente codificados no código xenético polos eucariotas. As estruturas mostradas son as estándar, non as formas ionizadas zwitteriónicas que aparecen cando están en disolución acuosa.
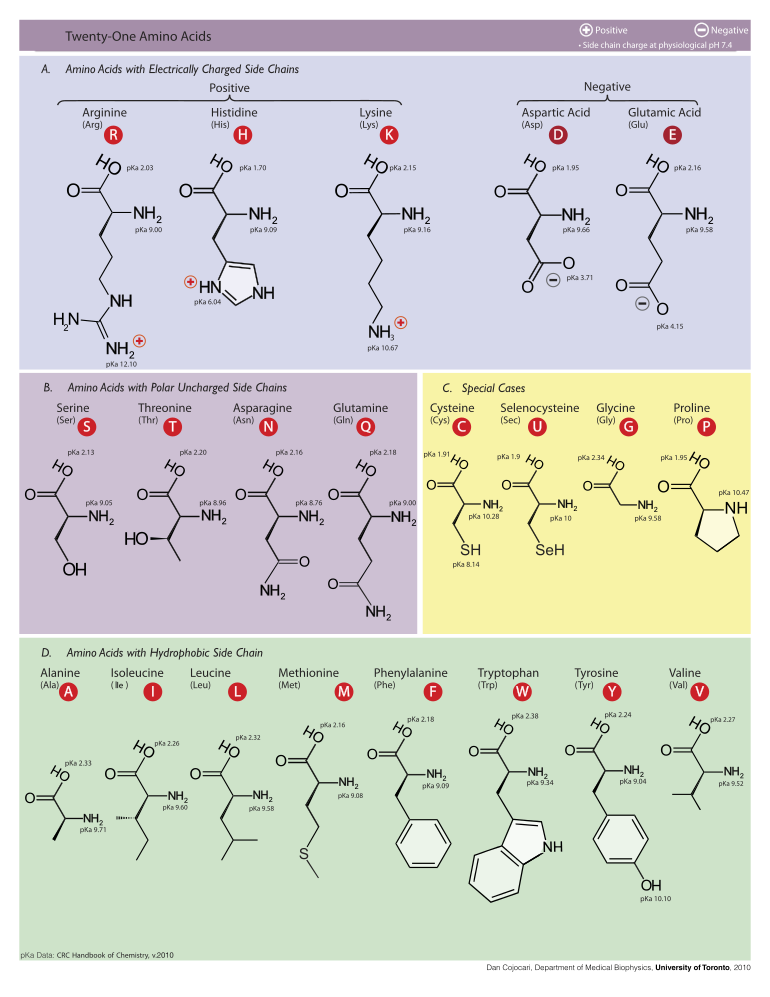
- L-Alanina
(Ala / A) - L-Arxinina
(Arg / R) - L-Asparaxina
(Asn / N) - Ácido L-aspártico
(Asp / D) - L-Cisteína
(Cys / C) - Ácido L-glutámico
(Glu / E) - L-Glutamina
(Gln / Q) - Glicina
(Gly / G) - L-Histidina
(His / H) - L-Isoleucina
(Ile / I) - L-Leucina
(Leu / L) - L-Lisina
(Lys / K) - L-Metionina
(Met / M) - L-Fenilalanina
(Phe / F) - L-Prolina
(Pro / P) - L-Serina
(Ser / S) - L-Treonina
(Thr / T) - L-Triptófano
(Trp / W) - L-Tirosina
(Tyr / Y) - L-Valina
(Val / V)
IUPAC/IUBMB recomenda as seguintes abreviacións para os dous aminoácidos seguintes:
- L-Selenocisteína
(Sec / U) - L-Pirrolisina
(Pyl / O)
Abreviacións non específicas
editarÁs veces a identidade dun aminoácido non pode ser determinada de forma inambigua. Por exemplo, en ocasións con certas técnicas non se pode saber se un aminoácido é asparaxina ou ácido aspártico. Neses casos úsanse abreviacións nas que se inclúe un X, como nos seguintes casos:
- Asx (B) pode ser "asparaxina ou ácido aspártico", que só se diferencian nun grupo amino.
- Glx (Z) pode ser "ácido glutámico ou glutamina", que só se diferncian nun grupo amino.
- Xle (J) pode ser "leucina ou isoleucina", que só se diferencian na posición dun metilo.
Propiedades químicas
editarNa seguinte táboa inclúense as propiedades químicas das cadeas laterais dos aminoácidos estándar. En canto ás masas téñase en conta que cando o aminoácido está na proteína ten unha masa lixeiramente menor, porque coa formación do enlace peptídico despréndese unha molécula de auga formada con átomos do aminoácido.
Propiedades químicas xerais:
| Aminoácido | Abrev. 1 letra | Abrev. 3 letras | Masa media (Da) | Punto isoeléctrico (pI) | pK1 (α-COOH) | pK2 (α-+NH3) |
|---|---|---|---|---|---|---|
| Alanina | A | Ala | 89.09404 | 6.01 | 2.35 | 9.87 |
| Cisteína | C | Cys | 121.15404 | 5.05 | 1.92 | 10.70 |
| Ácido aspártico | D | Asp | 133.10384 | 2.85 | 1.99 | 9.90 |
| Ácido glutámico | E | Glu | 147.13074 | 3.15 | 2.10 | 9.47 |
| Fenilalanina | F | Phe | 165.19184 | 5.49 | 2.20 | 9.31 |
| Glicina | G | Gly | 75.06714 | 6.06 | 2.35 | 9.78 |
| Histidina | H | His | 155.15634 | 7.60 | 1.80 | 9.33 |
| Isoleucina | I | Ile | 131.17464 | 6.05 | 2.32 | 9.76 |
| Lisina | K | Lys | 146.18934 | 9.60 | 2.16 | 9.06 |
| Leucina | L | Leu | 131.17464 | 6.01 | 2.33 | 9.74 |
| Metionina | M | Met | 149.20784 | 5.74 | 2.13 | 9.28 |
| Asparaxina | N | Asn | 132.11904 | 5.41 | 2.14 | 8.72 |
| Pirrolisina | O | Pyl | ||||
| Prolina | P | Pro | 115.13194 | 6.30 | 1.95 | 10.64 |
| Glutamina | Q | Gln | 146.14594 | 5.65 | 2.17 | 9.13 |
| Arxinina | R | Arg | 174.20274 | 10.76 | 1.82 | 8.99 |
| Serina | S | Ser | 105.09344 | 5.68 | 2.19 | 9.21 |
| Treonina | T | Thr | 119.12034 | 5.60 | 2.09 | 9.10 |
| Selenocisteína | U | Sec | 168.053 | |||
| Valina | V | Val | 117.14784 | 6.00 | 2.39 | 9.74 |
| Triptófano | W | Trp | 204.22844 | 5.89 | 2.46 | 9.41 |
| Tirosina | Y | Tyr | 181.19124 | 5.64 | 2.20 | 9.21 |
Propiedades da cadea lateral
editar| Aminoácido | Abrev. 1 letra | Abrev. 3 letras | Cadea lateral | Hidro- fóbica | pKa | Polar | pH | Pequena | Diminuta | Aromática ou Alifática | volume de van der Waals |
|---|---|---|---|---|---|---|---|---|---|---|---|
| Alanina | A | Ala | -CH3 | X | - | - | - | X | X | - | 67 |
| Cisteína | C | Cys | -CH2SH | X | 8.18 | - | ácida | X | - | - | 86 |
| Ácido aspártico | D | Asp | -CH2COOH | - | 3.90 | X | ácida | X | - | - | 91 |
| Ácido glutámico | E | Glu | -CH2CH2COOH | - | 4.07 | X | ácida | - | - | - | 109 |
| Fenilalanina | F | Phe | -CH2C6H5 | X | - | - | - | - | - | Aromática | 135 |
| Glicina | G | Gly | -H | X | - | - | - | X | X | - | 48 |
| Histidina | H | His | -CH2-C3H3N2 | - | 6.04 | X | básica feble | - | - | Aromática | 118 |
| Isoleucina | I | Ile | -CH(CH3)CH2CH3 | X | - | - | - | - | - | Alifática | 124 |
| Lisina | K | Lys | -(CH2)4NH2 | - | 10.54 | X | básica | - | - | - | 135 |
| Leucina | L | Leu | -CH2CH(CH3)2 | X | - | - | - | - | - | Alifática | 124 |
| Metionina | M | Met | -CH2CH2SCH3 | X | - | - | - | - | - | - | 124 |
| Asparaxina | N | Asn | -CH2CONH2 | - | - | X | - | X | - | - | 96 |
| Pirrolisina | O | Pyl | |||||||||
| Prolina | P | Pro | -CH2CH2CH2- | X | - | - | - | X | - | - | 90 |
| Glutamina | Q | Gln | -CH2CH2CONH2 | - | - | X | - | - | - | - | 114 |
| Arxinina | R | Arg | -(CH2)3NH-C(NH)NH2 | - | 12.48 | X | básica forte | - | - | - | 148 |
| Serina | S | Ser | -CH2OH | - | - | X | - | X | X | - | 73 |
| Treonina | T | Thr | -CH(OH)CH3 | - | - | X | ácida feble | X | - | - | 93 |
| Selenocisteína | U | Sec | -CH2SeH | X | 5.73 | - | - | X | - | - | |
| Valina | V | Val | -CH(CH3)2 | X | - | - | - | X | - | Alifática | 105 |
| Triptófano | W | Trp | -CH2C8H6N | X | - | - | - | - | - | Aromática | 163 |
| Tirosina | Y | Tyr | -CH2-C6H4OH | - | 10.46 | X | - | - | - | Aromática | 141 |
Nota: Os valores de pKa dos aminoácidos son tipicamente un pouco diferentes cando o aminoácido está formando parte dunha proteína.
Expresión xénica e bioquímica
editar| Aminoácido | Abrev. 1 letra | Abrev. 3 letras | Codón(s) | Frecuencia nas proteínas humanas (%) | Esencial‡ en humanos |
|---|---|---|---|---|---|
| Alanina | A | Ala | GCU, GCC, GCA, GCG | 7.8 | - |
| Cisteína | C | Cys | UGU, UGC | 1.9 | Condicionalmente |
| Ácido aspártico | D | Asp | GAU, GAC | 5.3 | - |
| Ácido glutámico | E | Glu | GAA, GAG | 6.3 | Condicionalmente |
| Fenilalanina | F | Phe | UUU, UUC | 3.9 | Si |
| Glicina | G | Gly | GGU, GGC, GGA, GGG | 7.2 | Condicionalmente |
| Histidina | H | His | CAU, CAC | 2.3 | Si |
| Isoleucina | I | Ile | AUU, AUC, AUA | 5.3 | Si |
| Lisina | K | Lys | AAA, AAG | 5.9 | Si |
| Leucina | L | Leu | UUA, UUG, CUU, CUC, CUA, CUG | 9.1 | Si |
| Metionina | M | Met | AUG | 2.3 | Si |
| Asparaxina | N | Asn | AAU, AAC | 4.3 | - |
| Pirrolisina | O | Pyl | UAG* | - | |
| Prolina | P | Pro | CCU, CCC, CCA, CCG | 5.2 | - |
| Glutamina | Q | Gln | CAA, CAG | 4.2 | - |
| Arxinina | R | Arg | CGU, CGC, CGA, CGG, AGA, AGG | 5.1 | Condicionalmente |
| Serina | S | Ser | UCU, UCC, UCA, UCG, AGU, AGC | 6.8 | - |
| Treonina | T | Thr | ACU, ACC, ACA, ACG | 5.9 | Si |
| Selenocisteína | U | Sec | UGA** | - | |
| Valina | V | Val | GUU, GUC, GUA, GUG | 6.6 | Si |
| Triptófano | W | Trp | UGG | 1.4 | Si |
| Tirosina | Y | Tyr | UAU, UAC | 3.2 | Condicionalmente |
| Codón de stop † | - | Term | UAA, UAG, UGA | - | - |
* UAG é normalmente o codón ámbar de stop, pero codifica a pirrolisina se está presente un elemento PYLIS e un ARNt especial.
** UGA é normalmente o codón de stop ópalo, pero codifica selenocisteína se está presente un elemento SECIS.
† O codón de stop non é un aminoácido, pero inclúese para completar os codóns do código.
‡ Un aminoácido esencial non pode ser sintetizado polos humanos e debe tomarse na dieta. Os aminoácidos condicionalmente esenciais non é imprescindible tomalos na dieta normalmente, pero si en determinados individuos en certas condicións.
Espectrometría de masas
editarNa espectrometría de masas dos péptidos e proteínas, é útil coñecer as masas dos residuos de aminoácidos. A masa do péptido ou proteína é a suma das masas dos residuos que a forman máis unha molécula de auga (a que se perdeu durante a formación do enlace peptídico).[6]
| Aminoácido | Abrev. 1 letra | Abrev. 3 letras | Fórmula | Masa monoisotópica (Da) | Masa media (Da) |
|---|---|---|---|---|---|
| Alanina | A | Ala | C3H5NO | 71.03711 | 71.0788 |
| Cisteína | C | Cys | C3H5NOS | 103.00919 | 103.1388 |
| Ácido aspártico | D | Asp | C4H5NO3 | 115.02694 | 115.0886 |
| Ácido glutámico | E | Glu | C5H7NO3 | 129.04259 | 129.1155 |
| Fenilalanina | F | Phe | C9H9NO | 147.06841 | 147.1766 |
| Glicina | G | Gly | C2H3NO | 57.02146 | 57.0519 |
| Histidina | H | His | C6H7N3O | 137.05891 | 137.1411 |
| Isoleucina | I | Ile | C6H11NO | 113.08406 | 113.1594 |
| Lisina | K | Lys | C6H12N2O | 128.09496 | 128.1741 |
| Leucina | L | Leu | C6H11NO | 113.08406 | 113.1594 |
| Metionina | M | Met | C5H9NOS | 131.04049 | 131.1986 |
| Asparaxina | N | Asn | C4H6N2O2 | 114.04293 | 114.1039 |
| Pirrolisina | O | Pyl | C12H21N3O3 | 255.15829 | 255.3172 |
| Prolina | P | Pro | C5H7NO | 97.05276 | 97.1167 |
| Glutamina | Q | Gln | C5H8N2O2 | 128.05858 | 128.1307 |
| Arxinina | R | Arg | C6H12N4O | 156.10111 | 156.1875 |
| Serina | S | Ser | C3H5NO2 | 87.03203 | 87.0782 |
| Treonina | T | Thr | C4H7NO2 | 101.04768 | 101.1051 |
| Selenocisteína | U | Sec | C3H5NOSe | 150.95364 | 150.0388 |
| Valina | V | Val | C5H9NO | 99.06841 | 99.1326 |
| Triptófano | W | Trp | C11H10N2O | 186.07931 | 186.2132 |
| Tirosina | Y | Tyr | C9H9NO2 | 163.06333 | 163.1760 |
Estequiometría e custo metabólico
editarA seguinte táboa indica a abundancia de aminoácidos na bacteria E. coli e o custo metabólico en ATP da síntese dos aminoácidos. Os números negativos indican que o proceso metabólico é enerxeticamente favorable e non ten un custo neto de ATP para a célula.[7] Para calcular a abundancia dos aminoácidos incluíronse as molécuas libres e as polimerizadas nas proteínas.
| Aminoácido | Abundancia (# de moléculas (×108) por célula de Escherichia coli) | custo de ATP na síntese en condicións aeróbicas | custo de ATP na síntese en condicións anaeróbicas |
|---|---|---|---|
| Alanina | 2.9 | -1 | 1 |
| Cisteína | 0.52 | 11 | 15 |
| Ácido aspártico | 1.4 | 0 | 2 |
| Ácido glutámico | 1.5 | -7 | -1 |
| Fenilalanina | 1.1 | -6 | 2 |
| Glicina | 3.5 | -2 | 2 |
| Histidina | 0.54 | 1 | 7 |
| Isoleucina | 1.7 | 7 | 11 |
| Lisina | 2.0 | 5 | 9 |
| Leucina | 2.6 | -9 | 1 |
| Metionina | 0.88 | 21 | 23 |
| Asparaxina | 1.4 | 3 | 5 |
| Prolina | 1.3 | -2 | 4 |
| Glutamina | 1.5 | -6 | 0 |
| Arxinina | 1.7 | 5 | 13 |
| Serina | 1.2 | -2 | 2 |
| Treonina | 1.5 | 6 | 8 |
| Triptófano | 0.33 | -7 | 7 |
| Tirosina | 0.79 | -8 | 2 |
| Valina | 2.4 | -2 | 2 |
Catabolismo
editarOs aminoácidos poden clasificarse de acordo coas posibilidades metabólicas dos produtos finais do seu catabolismo en tres grupos:[8]
- Glicoxénicos. Os produtos finais poden entrar nas vías metabólicas da gliconeoxénese que orixinan glicosa.
- Cetoxénicos. Os produtos finais non poden orixinar glicosa, pero si poden orixinar corpos cetónicos e lípidos.
- Aminoácidos que poden sintetizar ambas as cousas, polo que son á vez glicoxénicos e cetoxénicos.

Notas
editar- ↑ Ambrogelly A, Palioura S, Söll D (2007). "Natural expansion of the genetic code". Nat Chem Biol 3 (1): 29–35. PMID 17173027. doi:10.1038/nchembio847.
- ↑ Théobald-Dietrich A, Giegé R, Rudinger-Thirion J (2005). "Evidence for the existence in mRNAs of a hairpin element responsible for ribosome dependent pyrrolysine insertion into proteins". Biochimie 87 (9-10): 813–7. PMID 16164991. doi:10.1016/j.biochi.2005.03.006.
- ↑ Zhang Y, Baranov PV, Atkins JF, Gladyshev VN (2005). "Pyrrolysine and selenocysteine use dissimilar decoding strategies". J. Biol. Chem. 280 (21): 20740–51. PMID 15788401. doi:10.1074/jbc.M501458200.
- ↑ Michael Rother, Joseph A. Krzycki. Selenocysteine, Pyrrolysine, and the Unique Energy Metabolism of Methanogenic Archaea. Archaea. 2010; 2010: 453642.Published online 2010 August 17. doi: 10.1155/2010/453642 PMCID: PMC2933860. [1]
- ↑ Erives A (2011). "A Model of Proto-Anti-Codon RNA Enzymes Requiring L-Amino Acid Homochirality". J Molecular Evolution 73: 10–22. PMID 21779963. doi:10.1007/s00239-011-9453-4.
- ↑ "The amino acid masses". ExPASy. Consultado o 2009-01-06.
- ↑ Physical Biology of the Cell (Garland Science) p. 178
- ↑ Chapter 20 (Amino Acid Degradation and Synthesis) in: Denise R., PhD. Ferrier. Lippincott Williams & Wilkins, ed. Lippincott's Illustrated Reviews: Biochemistry (Lippincott's Illustrated Reviews). Hagerstwon, MD. ISBN 0-7817-2265-9.
Véxase tamén
editarBibliografía
editar- Nelson David L., Cox, Michael M. (2000). Worth Publishers, ed. Lehninger Principles of Biochemistry (3rd ed.). ISBN 1-57259-153-6.
- Kyte J., Doolittle, R. F. (1982). "A simple method for displaying the hydropathic character of a protein". J. Mol. Biol. 157 (1): 105–132. PMID 7108955. doi:10.1016/0022-2836(82)90515-0.
- Meierhenrich Uwe J. (2008). Springer, ed. Amino acids and the asymmetry of life (1st ed.). ISBN 978-3-540-76885-2.